最近在做突变分析,偶然看到有myVCF这个小工具,但是搜到的资料实在有限,自己摸索了摸索,将安装及使用过程总结一下。
安装
myVCF的功能,简单一句话,就是可视化vcf文件。我用到的就是这块。myVCF下载路径,在GitHub上。
推荐安装到Linux系统上,因为我在Windows上安装折腾了很久也没成功。
- 克隆GitHub上myVCF的源码。例如我安装到以下目录。
cd /home/username/software
git clone https://github.com/apietrelli/myVCF.git
- 在Linux系统下自己的home目录下创建python2环境,并安装依赖包。
cd /home/username/envis
virtualenv -p /usr/bin/python2 python27
pip install -r /home/username/software/myVCF-master/requirements.txt
使用
- 用终端调用时,我这里使用Xmanager Enterprise 5,因为这个里面的Xshell可以转发到X11桌面,不然的话无法显示,会报错:no display name and no $DISPLAY environment variable。
/home/username/envis/python27/bin/python2.7 -u /home/username/software/myVCF-master/myVCF_GUI.py
调用成功会显示如图界面,点击“Run myVCF”即可弹出火狐浏览器,进入软件界面。
2. 进入界面后如下图所示。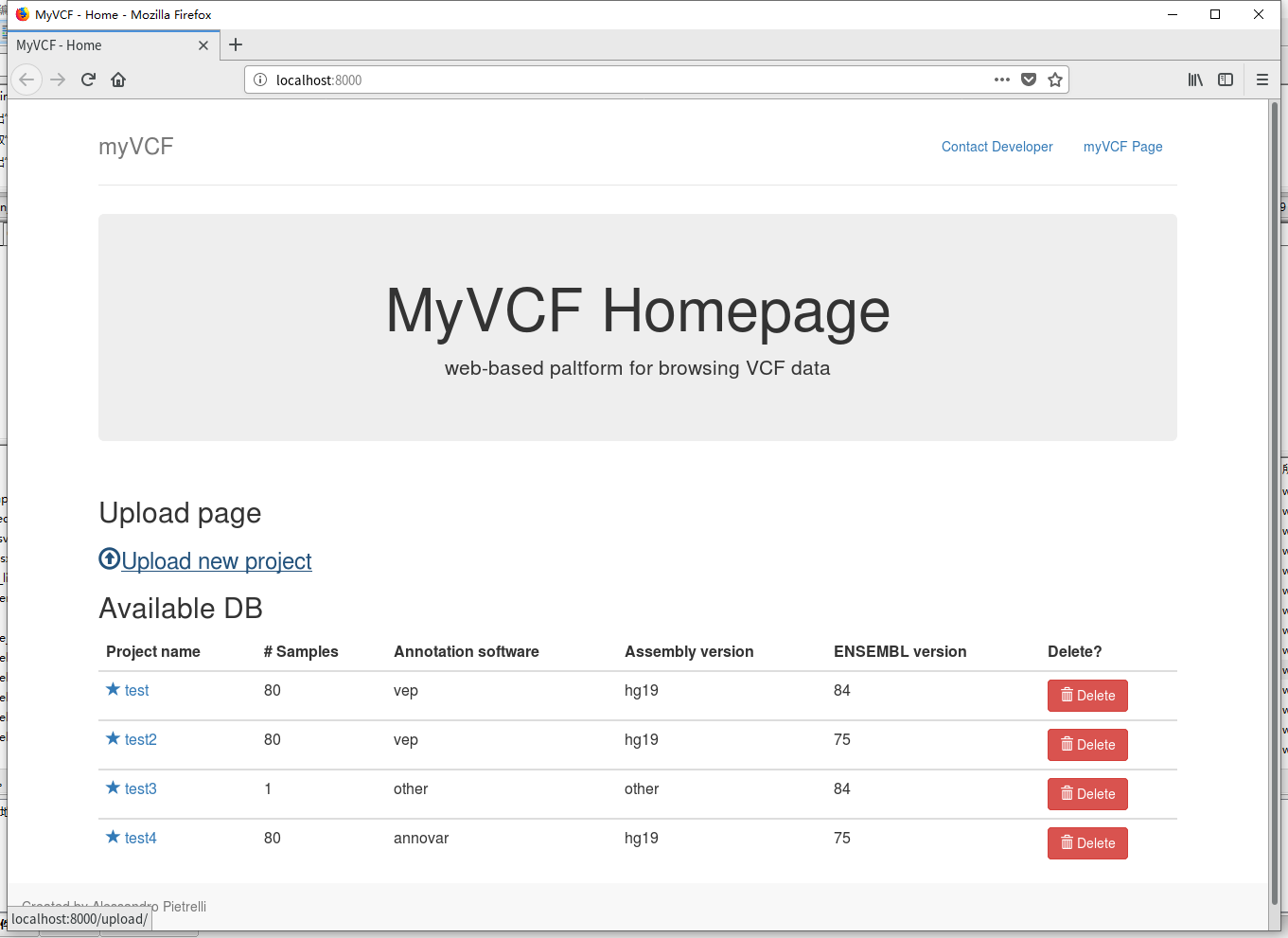 点击“Upload new project”即可创建自己的项目。软件自带几个例子,可以自己运行一下。要运行自己的数据需要将自己的vcf文件放入软件目录下:/home/username/software/myVCF-master/data/VCFs。
点击“Upload new project”即可创建自己的项目。软件自带几个例子,可以自己运行一下。要运行自己的数据需要将自己的vcf文件放入软件目录下:/home/username/software/myVCF-master/data/VCFs。
3. 运行了一个例子,结果展示如下,一系列可视化图片,简约美观,还能打印输出,能缩放,是交互式的,感觉很有趣。
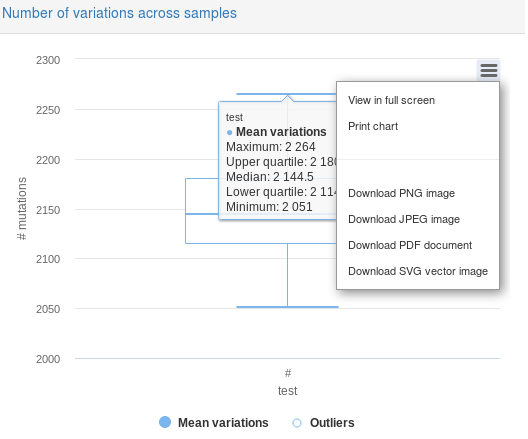
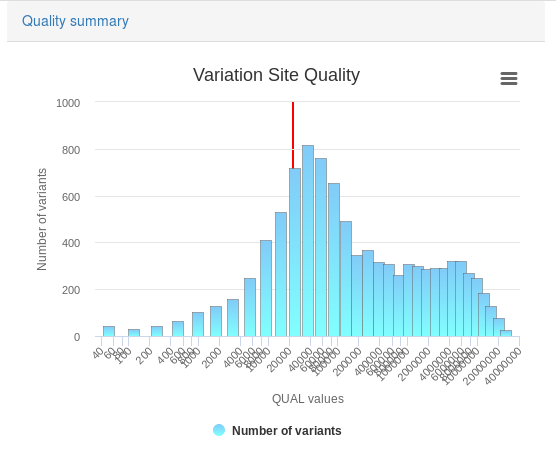
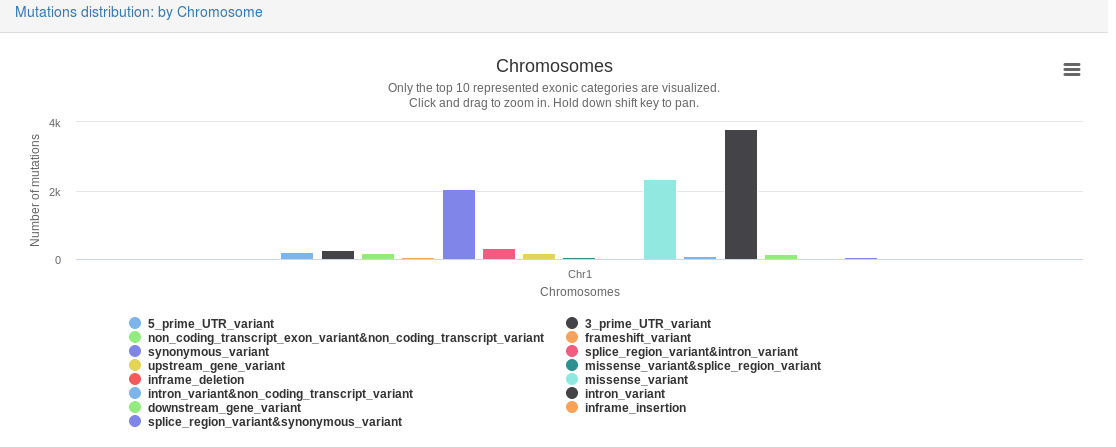
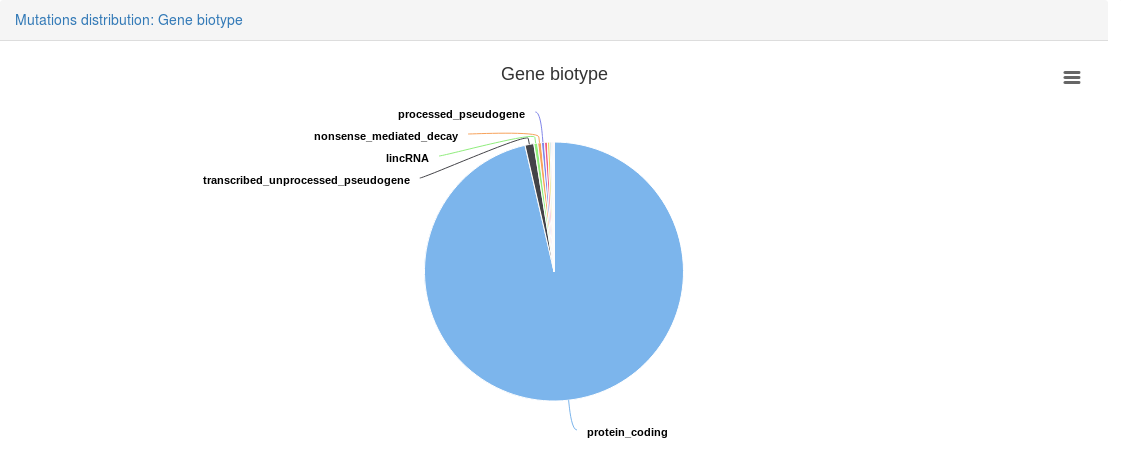
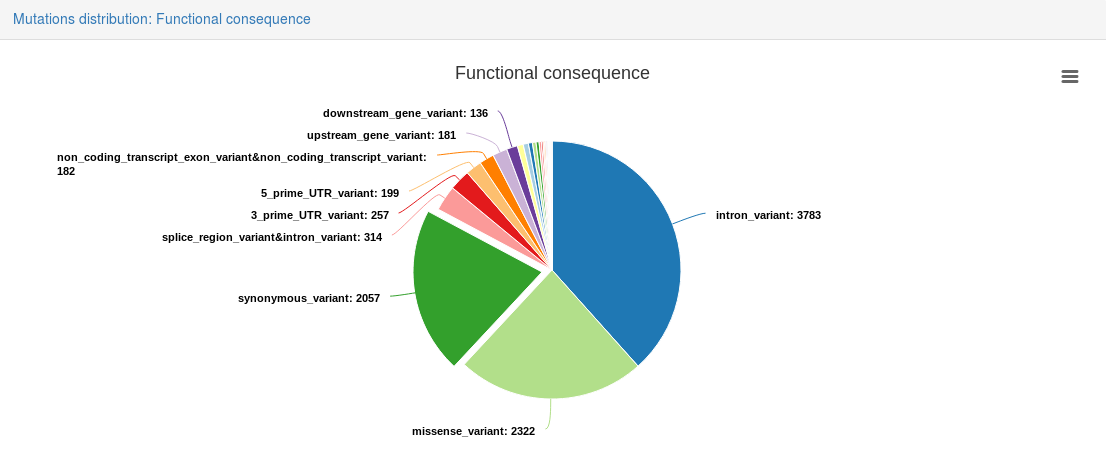
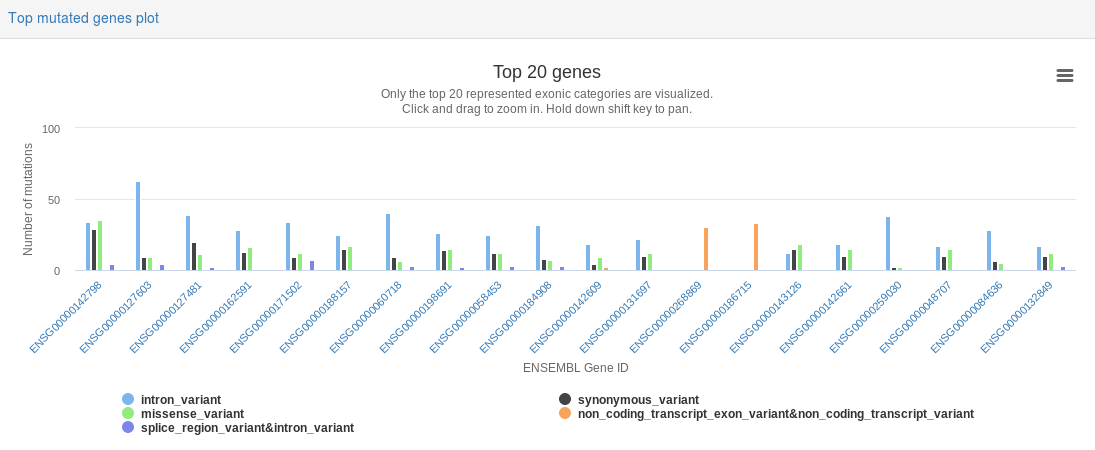
总结
测试了一些例子,我发现这个工具对注释后的vcf文件比较友好,没有注释的在可视化的步骤会受到影响。
挺有趣的小工具,但是也没有维护了。还有我运行的例子gene名称用的是ensembl ID,可能跟注释有关吧,看着不太方便。我工作中常用的是HGNC symbol。
跑了一个自己的未注释的vcf文件,结果只能出Mutations distribution: by Chromosome这张图,别的都没有。vcf注释工具我还没有研究,只能先这样了。